

蛋白质糖基化修饰的精准分析对于疾病机制研究、生物标志物发现,以及药物和疫苗开发至关重要。完整糖肽分析能获得特定糖基化位点与糖链的对应关系信息,是现代糖蛋白质组学研究中十分重要而极具挑战性的组成部分。目前,糖肽分析最常用的质谱采集模式是数据依赖性采集(DDA),存在母离子选择随机性的问题,其结果中存在较多的缺失值。近年来出现的数据非依赖性采集(DIA)模式能弥补DDA的缺点,已开始初步应用于糖蛋白质组学领域。然而,现有的糖肽DIA分析方法缺乏可靠的质控,难以处理多种糖肽在隔离窗口中共碎裂的复杂情况。
近日,复旦大学化学系乔亮课题组与合作者在《自然-通讯》(Nature Communications)期刊在线发表了题为“GproDIA enables data-independent acquisition glycoproteomics with comprehensive statistical control”的研究论文。该研究开发了完整糖肽DIA数据分析工具GproDIA,将以肽段为中心DIA分析的概念扩展到完整糖肽分析,通过二维错误发现率(FDR)和糖型推断算法提供全面的统计质控,实现糖肽的精准鉴定和定量。
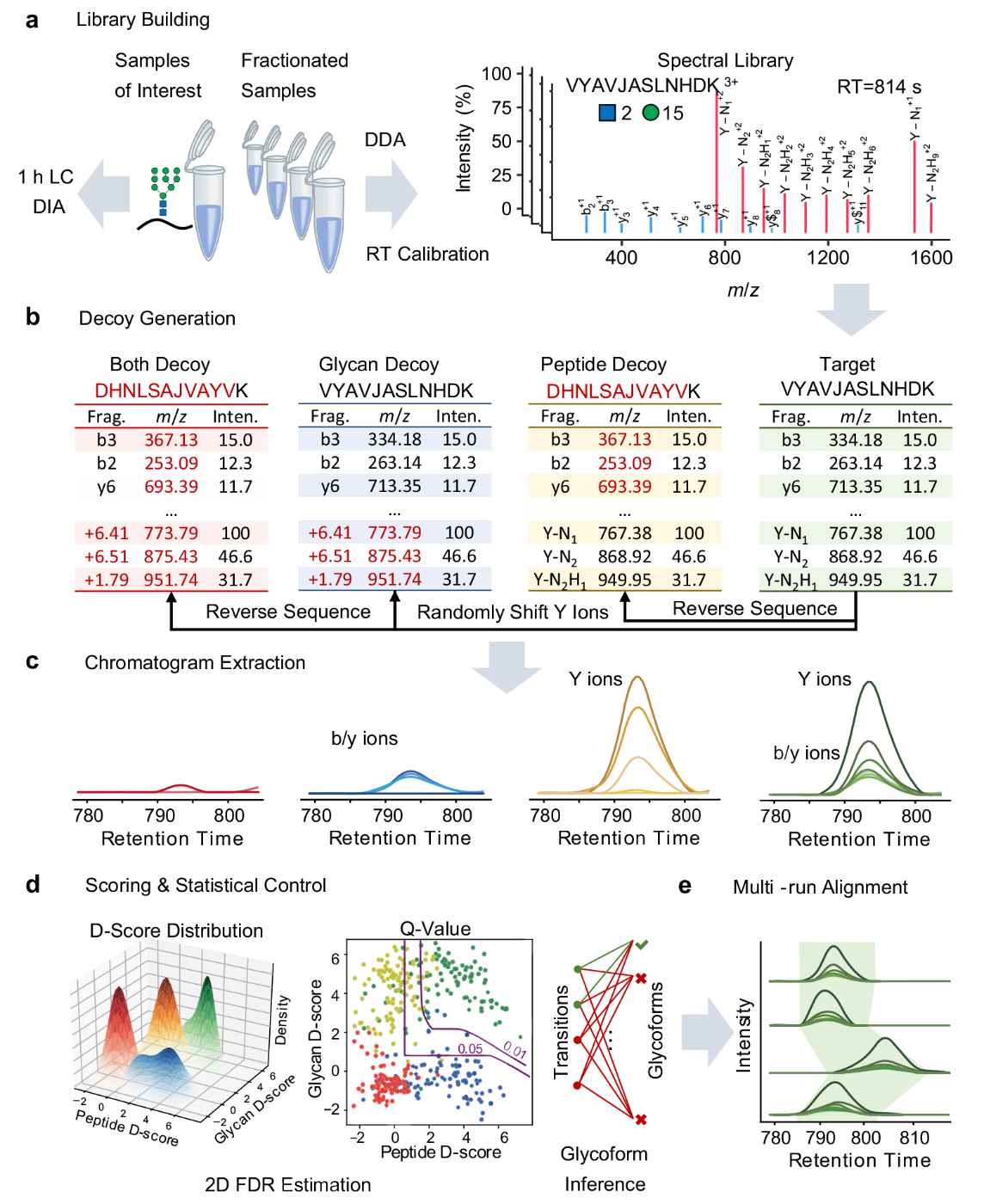
二维FDR算法首先为谱图库中目标糖肽生成肽段诱饵、糖链诱饵和糖肽双诱饵,从DIA数据中提取目标糖肽和三种类型诱饵的谱图信息并对肽段和糖链部分分别进行评分。然后采用双变量四组分混合模型拟合目标糖肽和诱饵的得分分布,由此估计每个目标糖肽匹配结果中肽段、糖链部分错误或两者都错误的概率(即局部FDR)和所报告全部结果的整体错误率(即全局FDR)。
对于复杂样品,糖肽的鉴定会受到隔离窗口中相同肽段序列上的其他糖型的潜在干扰。为此,研究人员进一步提出了一种糖型推断算法。该算法首先为DIA隔离窗口内的潜在糖型生成鉴定离子,从DIA数据中提取这些鉴定离子和目标糖肽母离子的谱图信息进行评分,然后采用贝叶斯层次模型将目标糖肽原先的得分同母离子和鉴定离子的得分加以整合,得到糖型的得分,从而对糖型的错误率进行质控。
研究人员将GproDIA在酵母和人血清N-糖肽样品的DIA数据上进行测试。结果表明,GproDIA在糖肽鉴定数、数据完整性,以及定量的准确性和精密度方面均优于现有基于DDA的方法。与现有的糖肽DIA分析方法相比,GproDIA不仅能排除肽段部分的错误鉴定结果,还能对糖链部分进行良好的质控,实现了更准确的糖肽鉴定。GproDIA为糖蛋白质组分析提供了一项有力工具,有望促进蛋白质糖基化相关疾病机制和生物标志物的研究。
据悉,基于DIA的蛋白质组学是乔亮研究员团队的研究兴趣之一,上述工作是该团队继2020年初在《自然-通讯》上发表研究论文报道基于深度学习预测谱图库的DIA分析方法(Nature Communications, 2020, 11: 146)之后,在DIA蛋白质组学研究中取得的又一重要成果。
化学系2017级直博生杨奕和复旦大学生物医学研究院曹纬倩青年副研究员为本文共同第一作者,乔亮研究员和曹纬倩为共同通讯作者。杨芃原教授对本研究给予了大力支持。该工作得到了国家自然科学基金和国家重点研发计划项目的资助。
全文链接:https://doi.org/10.1038/s41467-021-26246-3





