

蛋白质糖基化对细胞信号传导、免疫识别等生理功能至关重要。相比于其他翻译后修饰,糖基化修饰更加复杂,不但存在修饰位点的异质性,同一位点上的修饰基团还可能由不同的单糖按照不同的连接结构组成,具有高度结构异质性。完整糖肽分析需要实现肽段序列、糖基化位点和糖链结构的同时分析,对分析技术提出了极大挑战。
近日,复旦大学化学系乔亮团队将深度学习与糖蛋白质组学相结合,开发了DeepGP深度学习框架。DeepGP在糖肽串联质谱(MS/MS)分析方面取得了突破性进展。相关成果以“Deep learning prediction of glycopeptide tandem mass spectra powers glycoproteomics”为题,在《Nature Machine Intelligence》上发表。
糖蛋白质组研究通常采用色谱质谱联用对糖基化肽段的色谱保留时间、精确分子量和二级谱图进行高通量信息采集,之后结合精确分子量与二级谱图完成糖肽结构鉴定。传统的数据分析手段往往采用序列搜库的策略,基于碎片离子的质荷比信息判断潜在的糖基化修饰结构。这一策略的弊端在于当二级谱图中碎片离子不够丰富时则很难对相近的糖结构进行区分。
为了应对这一挑战,复旦大学团队开发了一个名为DeepGP的混合深度学习框架。DeepGP能够准确预测糖肽的二级谱图和色谱保留时间。得益于DeepGP的准确预测,可以利用完整二级谱图相似性比对来区分不同糖型,从而更加充分的挖掘谱图信息。
DeepGP结合了两种人工智能模型:一种擅长理解序列(Transformer),另一种擅长处理图形(图神经网络)。这种结合可以完美地处理糖基化肽段中的线性肽段序列和二维糖链结构。此外,研究团队还采用了预训练策略以克服糖蛋白质组学数据的稀缺性。
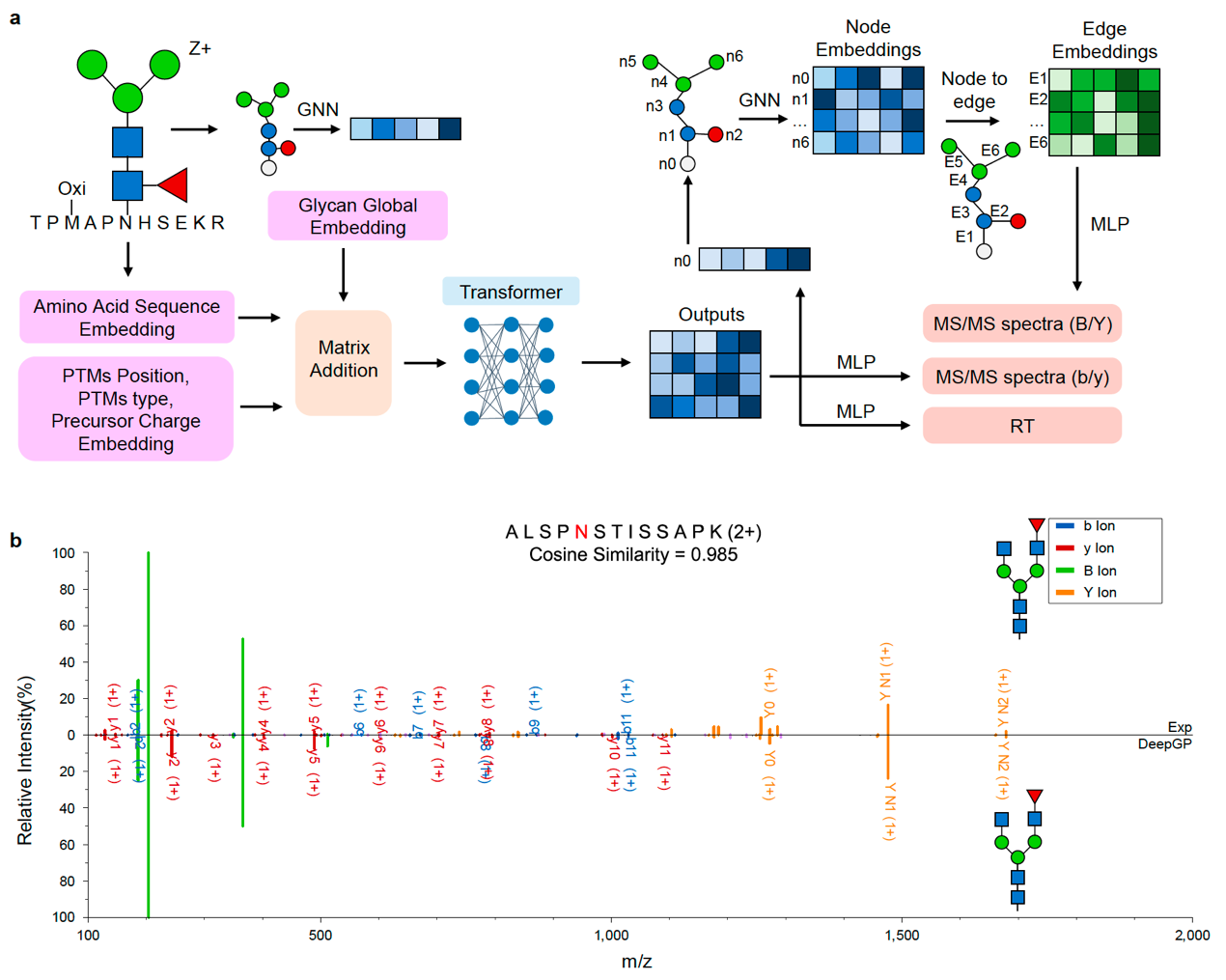
图1:(a) DeepGP模型结构示意图;(b) DeepGP预测的糖基化肽段二级谱图与实验二级谱图比对。(来源于发表论文)
通过在合成和实际数据集上的广泛测试,DeepGP已经被证明可以精准预测糖基化肽段的质谱二级谱图。预测谱图与实验谱图之间的相似性可以达到实验重复之间的相似性。基于DeepGP预测的二级谱图,可以在缺乏诊断离子的情况下对异构体糖肽进行区分。通过将DeepGP预测的二级谱图相似性打分与传统搜库得分进行整合,研究人员显著提升了糖基化肽段的鉴定灵敏度。在酵母和小鼠器官等不同的生物样本上进行测试,结合不同的诱饵陷阱库策略,研究团队证明DeepGP对糖基化肽段鉴定量提升达30%到170%,极大提升了蛋白质糖基化鉴定灵敏度。
DeepGP的开发是深度学习在生命分析领域的又一次成功应用。这项工作不仅推动了糖蛋白质组学技术的发展,也为未来的生物医学研究提供了强大的工具。随着技术的不断进步和应用的拓展,我们有理由相信,深度学习将在生物医学领域发挥越来越重要的作用。
复旦大学化学系博士生宗宇为论文第一作者,复旦大学计算机系博士生汪燠欣,邱锡鹏教授,黄萱菁教授对本文做出重要贡献,复旦大学化学系乔亮研究员为论文通讯作者。论文受到了复旦大学AI for Science等项目的资助。
全文链接:https://www.nature.com/articles/s42256-024-00875-x





